Un mapa 3D revela la organización del ADN en las células de la retina humana
«Se trata de la primera integración detallada de la topología del genoma regulador de la retina con las variantes genéticas asociadas a la degeneración macular asociada a la edad (DMAE) y al glaucoma, dos de las principales causas de pérdida de visión y ceguera», afirmó el investigador principal del estudio, el doctor Anand Swaroop, investigador principal y jefe del Laboratorio de Neurobiología, Neurodegeneración y Reparación del Instituto Nacional del Ojo (NEI, por sus siglas en inglés), que forma parte de los Institutos Nacionales de Salud.
Los científicos del Instituto Nacional del Ojo explican cómo la arquitectura genética determina la expresión de los genes, la función específica de los tejidos y el fenotipo de las enfermedades que provocan ceguera.
Las células de la retina humana adulta son neuronas sensoriales altamente especializadas que no se dividen, por lo que son relativamente estables para explorar cómo la estructura tridimensional de la cromatina contribuye a la expresión de la información genética.
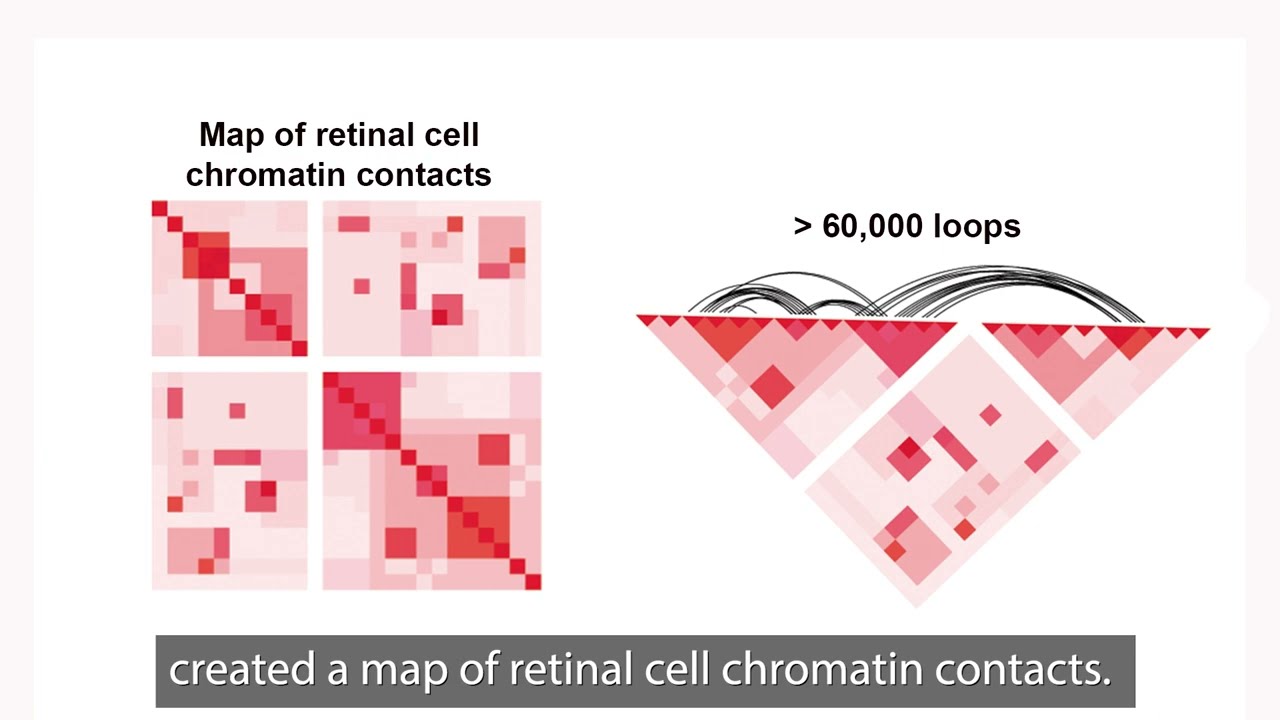
Los investigadores del Instituto Nacional del Ojo trazaron un mapa de la organización de la cromatina de las células de la retina humana, las fibras que empaquetan 3.000 millones de moléculas de ADN con longitud de nucleótidos en estructuras compactas que encajan en los cromosomas dentro del núcleo de cada célula. La red reguladora de genes resultante proporciona información sobre la regulación de la expresión génica en general, y de la función retiniana, tanto en las enfermedades oculares raras como en las comunes. El estudio se publica en Nature Communications.
Las células de la retina humana adulta son neuronas sensoriales altamente especializadas que no se dividen, por lo que son relativamente estables para explorar cómo la estructura tridimensional de la cromatina contribuye a la expresión de la información genética.
Las fibras de cromatina empaquetan largas hebras de ADN, que se enrollan alrededor de las proteínas histónicas y luego forman repetidos bucles para formar estructuras muy compactas. Todos esos bucles crean múltiples puntos de contacto en los que las secuencias genéticas que codifican proteínas interactúan con las secuencias reguladoras de los genes, como los superpotenciadores, los promotores y los factores de transcripción.
Esas secuencias no codificantes se consideraron durante mucho tiempo «ADN basura». Pero estudios más avanzados demuestran las formas en que estas secuencias controlan qué genes se transcriben y cuándo, arrojando luz sobre los mecanismos específicos por los que los elementos reguladores no codificantes ejercen el control incluso cuando su ubicación en una cadena de ADN está alejada de los genes que regulan.
Mediante la secuenciación profunda Hi-C, una herramienta utilizada para estudiar la organización del genoma en 3D, los investigadores crearon un mapa de alta resolución que incluía 704 millones de puntos de contacto dentro de la cromatina de las células de la retina. Los mapas se construyeron con muestras de retina post mortem de cuatro donantes humanos.
A continuación, los investigadores integraron ese mapa de topología de la cromatina con conjuntos de datos sobre genes y elementos reguladores de la retina. Lo que surgió fue una imagen dinámica de las interacciones dentro de la cromatina a lo largo del tiempo, incluidos los puntos calientes de la actividad genética y las áreas con diferentes grados de aislamiento de otras regiones del ADN.
Descubrieron distintos patrones de interacción en los genes de la retina, lo que sugiere que la organización tridimensional de la cromatina desempeña un papel importante en la regulación génica específica de los tejidos.
«Disponer de una imagen de tan alta resolución de la arquitectura genómica seguirá proporcionando información sobre el control genético de las funciones específicas de los tejidos», afirma Swaroop.
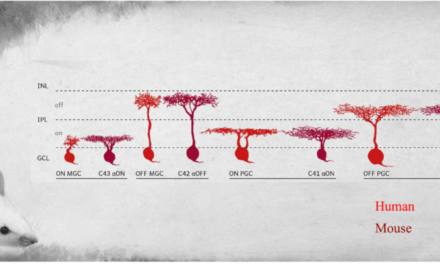
Además, las similitudes entre la organización de la cromatina de los ratones y la de los humanos sugieren su conservación en todas las especies, lo que subraya la importancia de los patrones de organización de la cromatina para la regulación de los genes de la retina. Más de un tercio (35,7%) de los pares de genes que interactúan a través de un bucle de cromatina en los ratones también lo hacen en la retina humana.
Los investigadores integraron el mapa topológico de la cromatina con datos sobre variantes genéticas identificadas a partir de estudios de asociación de todo el genoma por su implicación en la DMAE y el glaucoma, dos de las principales causas de pérdida de visión y ceguera. Los hallazgos apuntan a genes causales candidatos específicos implicados en esas enfermedades.
El mapa regulador del genoma integrado también ayudará a evaluar los genes asociados a otras enfermedades comunes de la retina, como la retinopatía diabética, a determinar la heredabilidad que falta y a comprender las correlaciones genotipo-fenotipo en las enfermedades hereditarias de la retina y la mácula.
Traducción: Asociación Mácula Retina
Fuente